摘要
背景
复发性阿弗他性口炎(RAS)经常影响患者的生活质量,因为长期持续和反复发作的灼痛。然而,目前有效的医疗疗法暂时很少。药物靶点识别是药物发现的第一步,通常是寻找潜在靶点候选分子与探针小分子之间的最佳相互作用模式。因此,阐明RAS发病的分子机制,探索RAS药物治疗的潜在分子靶点至关重要。
方法
应用生物信息学数据挖掘技术探索潜在的新靶点,利用加权基因共表达网络分析(WGCNA)构建GSE37265基因芯片数据的共表达模块,并利用分子复合物检测(MCODE)插件对中心基因进行识别。
结果
共鉴定出16个共表达模块,其中绿松石模块中鉴定出30个轮毂基因。此外,对感兴趣模块中的Hub基因进行功能分析,发现该Hub基因主要参与免疫应答、病毒感染、上皮细胞、信号转导等相关通路。在网络中确定了两个聚类(高度互联区域),得分分别为17.647和10,聚类1和聚类2由STAT1和ICAM1连接,推测STAT1可能是RAS的主基因。最后从tcsp数据库中筛选出染料木素、大豆苷元、山奈酚、白藜芦醇、迷迭香酸、雷公藤甲素、槲皮素和(-)-表没食子儿茶素-3-没食子酸酯,它们都是STAT-1抑制剂。反分子对接的结果表明,除了雷公藤甲素、(-)-表没食子儿茶素-3-没食子酸酯和白藜芦醇外,其他5种结构相似的化合物(类黄酮)可能与STAT1蛋白的同一位置结合,对接得分不同。
结论
我们的研究确定了STAT1作为可能有助于RAS诊断和潜在治疗靶点的潜在生物标志物,我们也可以从STAT-1抑制剂中筛选RAS治疗药物。
背景
复发性口腔炎(RAS)被认为是最常见的口腔黏膜疾病[1].RAS是一种疼痛的(包括前驱性烧灼感)、边界清晰的圆形溃疡,被黄灰色假膜覆盖,周围被红斑晕包围,或在整个口腔内形成更大的、平台状、不规则病变[2,3.].RAS的典型表现是复发性、自限性溃疡,主要影响非角化口腔黏膜(通常位于颊部、唇部黏膜、舌部和口腔底部)。严重角化的腭和牙龈粘膜受累较少[4].由于口腔疾病经常对语言、营养、外貌、自尊和社会交往产生有害影响,特别是RAS经常影响患者的生活质量,导致长时间持续和反复发作的灼痛[5].
虽然RAS发病的分子机制尚不清楚,但可能涉及免疫反应、慢性炎症、氧化应激、细胞外基质等生物学过程。[5,6,7].因此,阐明RAS发病的分子机制,探索RAS药物治疗的潜在分子靶点至关重要。随着基因芯片和高通量测序技术的广泛应用,与基因组相关的数据库积累了大量的数据[8].计算机化方法已应用于发现作为疾病潜在生物标志物的特征基因[9].如何利用生物信息学技术深入挖掘这些数据的潜在价值,已成为研究疾病分子机制的重要方向之一。生物信息学分析方法可以帮助我们从系统的角度研究疾病的分子机制,发现潜在的治疗靶点[8,9].在所有生物信息学分析方法中,加权基因共表达网络分析(WGCNA)是一种用于分析多样本基因表达模式的先进而全面的算法方法[10].WGCNA的独特优势是能够分析基因表达谱,通过具有共同生物关系或功能的相似行为基因聚类基因,形成共表达模块,揭示基因网络和信号通路,识别模内枢纽基因[11].它已被成功地用于研究各种生物过程,被证明对识别候选生物标志物和潜在治疗靶点非常有帮助[10,11].
RAS的管理取决于病变的频率和严重程度[5].大多数RAS病例可以通过局部治疗得到充分的治疗,目前的治疗方法包括缓解疼痛、抗炎、促进溃疡愈合,而主要包括抗生素治疗、激素治疗、药物漱口水和激光治疗[5,6,7].然而,目前治疗RAS的有效药物疗法暂时很少。中药积累了许多治疗疾病的天然药物,分子生物学和药物分子靶点识别技术在当前中草药研究中得到越来越广泛的应用[12].药物靶点识别是药物发现的第一步,包括许多不同的寻找基因和蛋白质的算法,靶点识别的问题通常是寻找潜在靶点候选分子与探针小分子之间的最佳相互作用模式[13].先导化合物的确定已经发展了许多计算机模拟分析技术,如基于结构的目标发现方法(如药效团、相似结合位点、基于指纹的相互作用方法、分子对接等),有代表性的数据库如tcsp、Pharmmapper等,计算并保存了大量天然活性化学成分的目标数据[12,13,14].
在本研究中,我们利用多种生物信息学分析工具对28例RAS患者样本的基因芯片数据进行了深入的数据挖掘,最终确定STAT1可能是影响RAS过程的关键靶点,同时也是潜在的治疗靶点,在此靶点的基础上筛选了8种草药的天然化学成分,可能成为RAS局部治疗的潜在药物,为后续研究提供了新的方向。
方法
数据集的收集和验证
用于我们分析的基因表达数据集是从基因表达综合(GEO)数据库(http://www.ncbi.nlm.nih.gov/geo/),以“复发性口腔炎”作为搜索关键词。筛选并下载了由Baccaglini L等人提供的GEO跟踪号GSE37265和平台入口号GPL570的数据集。样品收集和微阵列数据集由佛罗里达大学分子遗传学和微生物学微阵列实验室103完成。在这个数据集中,转录谱是从对照组的正常组织和患者的溃疡和非溃疡组织中建立的。用Affymetrix Human Genome U133 Plus 2.0 Array检测转录谱。
差异表达基因(DEGs)分析
利用微阵列平台数据表对矩阵文件进行正式基因符号标注,使用“sva”R包对原始表达数据进行批量归一化处理,得到归一化后的包含数据的基因表达矩阵文件,用于DEGs分析。使用“limma”R包进行DEGs分析。的P用t检验方法计算基因的-值,用Benjamini和Hochberg的方法计算调整值P价值。
构建mirna -基因相互作用网络
MicroRNA (miRNA)在哺乳动物的发育调控中起着关键作用。基于TarBase (http://www.microrna.gr/tarbase),是现有最大的人工策划靶数据库,从高通量实验中衍生出索引靶,提供数百万个高质量的人工策划实验验证的mirna -基因相互作用[15].
建设信令信息网络
生物体内基因和蛋白质表达的调控离不开信号分子等化学物质的广泛参与,信号信息网络构建基于SIGNOR 2.0 (https://signor.uniroma2.it/),这是一个公共资源库,它存储了参与信号转导关系的蛋白质和其他生物相关实体(化学物质、表型、复合物等)之间的手动注释因果关系,以图形的方式表示为有符号有向图[16].
加权基因共表达网络分析及共表达网络构建
利用典型的系统生物学算法R (3.6.2) WGCNA包进行加权基因相关网络分析,构建共表达网络。首先,我们对样本进行聚类分析,通过hclust函数[10,11].
基因模块的基因集富集分析
构建的模块由多个基因组成,然后对模块中的DEGs进行功能富集分析。为了获得这些模块所涉及的生物功能和信号通路,使用GSEA软件(GSEA版本4.0.3)对模块中的DEGs进行基因本体(GO)分析和(KEGG)通路分析[17].经过多次试验校准后,我们使用“调整”P以< 0.002”和“FDR < 0.05”为阈值识别富集术语,筛选出最重要的前10个术语。
PPI网络
构建了一个PPI网络来评估基因之间的相互作用,有助于我们探索新的分子机制。感兴趣的模块使用STRING 11.0 (https://string-db.org/),以搜寻不同蛋白质之间的相互作用[18].选取从WGCNA中获得的保存模块中共有的基因和一致性显著的DEGs构建PPI网络,使用Cytoscape 3.7.2软件进行可视化[19].在PPI网络中,一个节点代表一个基因;两个节点之间的无向链接是一条边,表示两个基因之间的相互作用;节点的程度与网络中某个基因与其他基因相互作用的数量相对应,只有经过实验验证的相互作用综合得分大于0.9才被选为显著性。以节点度和相互作用分数为关键拓扑参数,将最大连接基因非正式地称为枢纽基因。
轮毂基因的鉴定与验证
一个基因的模块内连通性等于该基因与该模块中其他基因的关联度之和。选择模块内连通性最高的前30个基因作为枢纽基因。筛选出感兴趣的基因模块后,利用Cytoscape构建加权基因共表达网络,利用分子复合物检测(MCODE)插件对枢纽基因进行识别。基因调控网络可以帮助我们准确筛选可能参与靶基因调控的候选基因,并可以利用已知基因的功能预测未知基因的功能。
对枢纽基因起作用的天然药物有效成分的筛选
tccmsp是一个专注于活性成分和靶点探索的中草药药理学平台,目前已收集到499种中草药,共12144种化学成分,以及天然化合物的药代动力学特性[20.].药物靶点从两个来源获得:(1)从HIT数据库中检索实验验证的药物靶点对(2)利用构建的SysDT模型预测化合物的潜在靶点[20.].为了从tccmsp数据库中获取相关成分,我们选择搜索类别为“靶标名称”,关键词设置为“转录1-alpha/beta的信号转换器和激活器”进行搜索,DL≥0.1为过滤条件。
预测潜在目标
PharmMapper [21]的设计目的是利用药效团映射方法识别给定探针小分子的潜在候选靶标。上传查询文件:。Mol2,参数设置:Generate conformer: Yes;最大生成构象:300;选择靶点集:可药物药效团模型(v2017, 16,159);保留匹配目标数(最大1000):500。在提交并等待计算完成后,结果将以csv文件格式保存。
反向分子对接验证
分子对接由AutoDock Vina完成[22].所有生物分子的可视化均由PyMol Software完成[23].
统计测试
按照生物学的惯例,P≤0.05被认为是统计显著性的截止点。
结果
数据集的验证
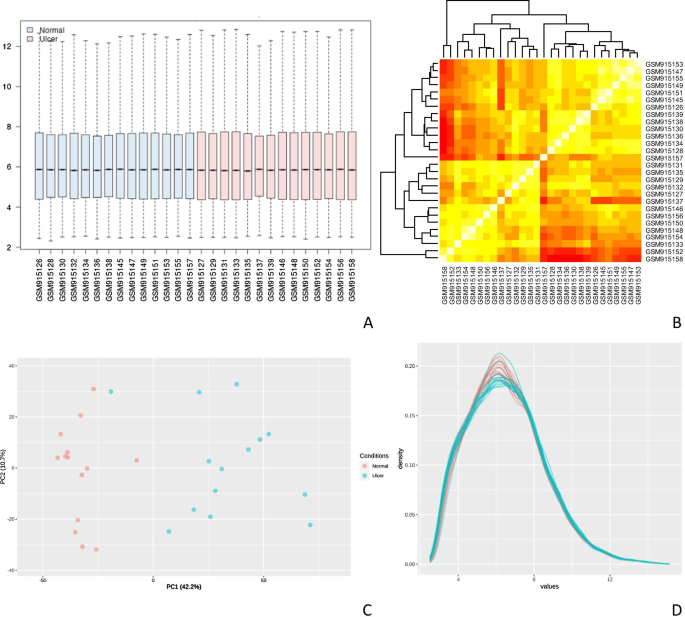
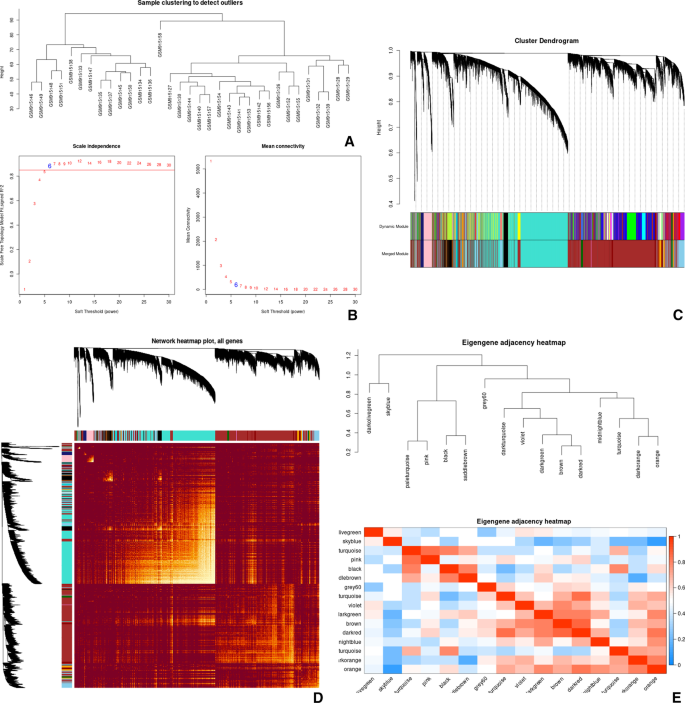
我们在分析之前对GSE37265的原始数据进行归一化处理,该箱线图显示了数据集中原始读取计数的分布(图1)。1A).为了进一步验证组内数据的可重复性,我们采用Pearson相关检验和主成分分析(PCA)。颜色反映了相关性的强弱,当0 <相关性< 1时,存在正相关性。−1 <相关性< 0时,存在负相关,数值绝对值越大,相关性越强,说明GSE37265数据集中健康组样本与RAS组样本之间存在很强的相关性(图1)。1A).基于PCA, GSE37265数据集的组内数据重复性是可以接受的。在PCA图中,分别以主成分1 (PC1)和主成分2 (PC2)为x轴和y轴绘制散点图,其中每个点代表一个样本,两个样本相距越远,两个样本在基因表达模式上的差异越大。对照组与复发性口腔炎组各样本间主成分-1 (PC1)维数均为可接受(图1)。1B).诊断图汇总了每个基因样本中reads的标准差与均值测量值,显示计数与方差之间的相关性是可以接受的。密度与读取计数log2的比值图显示了不同计数在健康组和RAS组的相对分布。
RAS与健康对照差异表达基因(DEGS)
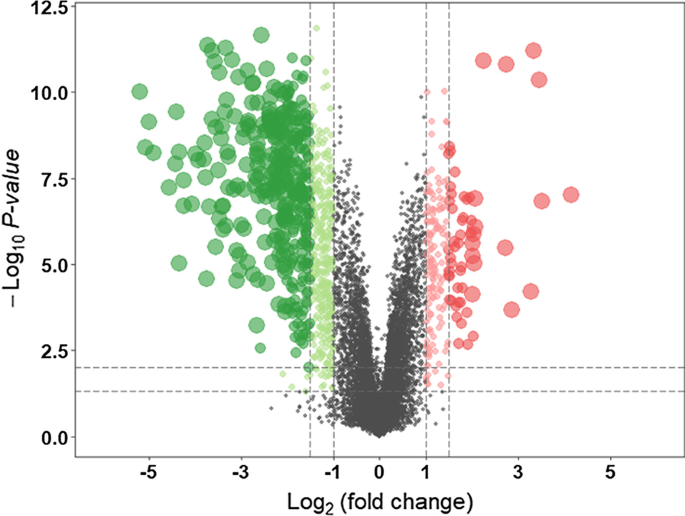
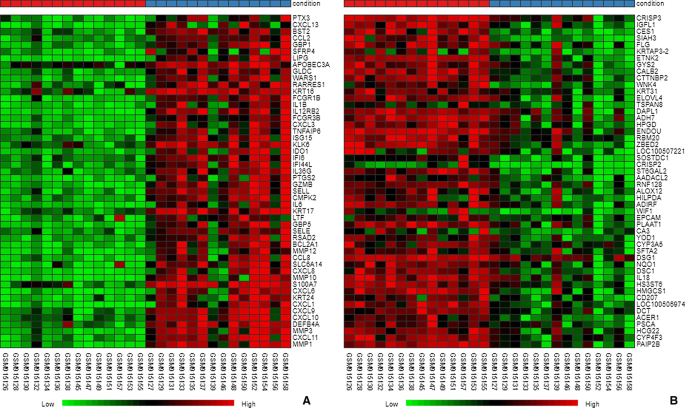
应用R软件及其扩展包对来自GSE37265队列的复发性口腔炎样本进行分析。数据预处理后得到基因表达矩阵(包含12548个基因)。共识别出187个deg,阈值为|log2 (fold-change) | > 2和P< 0.05,由125个下调基因和18个上调基因组成,所有探针的火山图如图所示。2.50个最显著的下调基因和上调基因通过热图可视化(图5)。3.).红色代表表达增加,而蓝色代表表达减少。RAS样本中上调最多的基因为DAPL1、TSPAN8、ELOVL4、KRT31、WNK4、CTTNBP2、CALB2、GYS2、ETNK2、KRTAP3-2,而下调最多的基因为MMP1、CXCL11、MMP3、DEFB4A、CXCL10、CXCL9、CXCL1、KRT24、CXCL6、S100A7、MMP10、CXCL8、SLC6A14、CCL8、MMP12。
构建mirna -基因相互作用网络
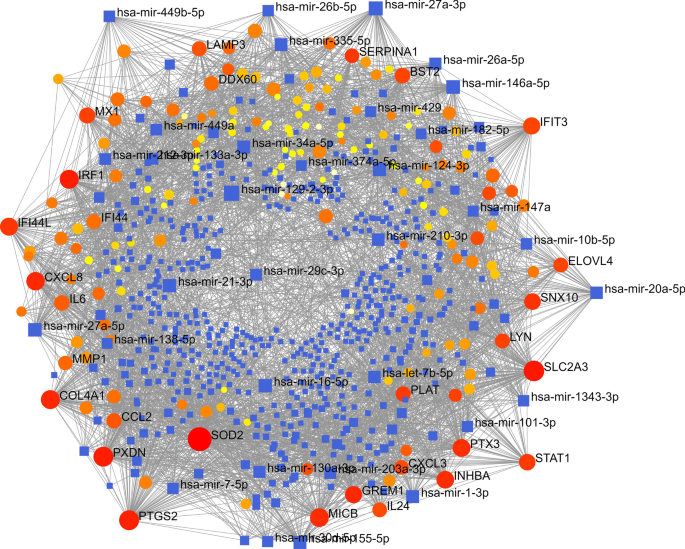
如图所示。4,我们基于TarBase, Table构建了DEGs的mirna -基因相互作用网络1根据相互作用程度列出了前20位高水平基因,揭示了SOD2、SLC2A3、PXDN、PTGS2、IRF1、COL4A1、MICB、CXCL8等可能在miRNA调控网络中发挥重要作用。
构建基于SIGNOR的信令信息网络
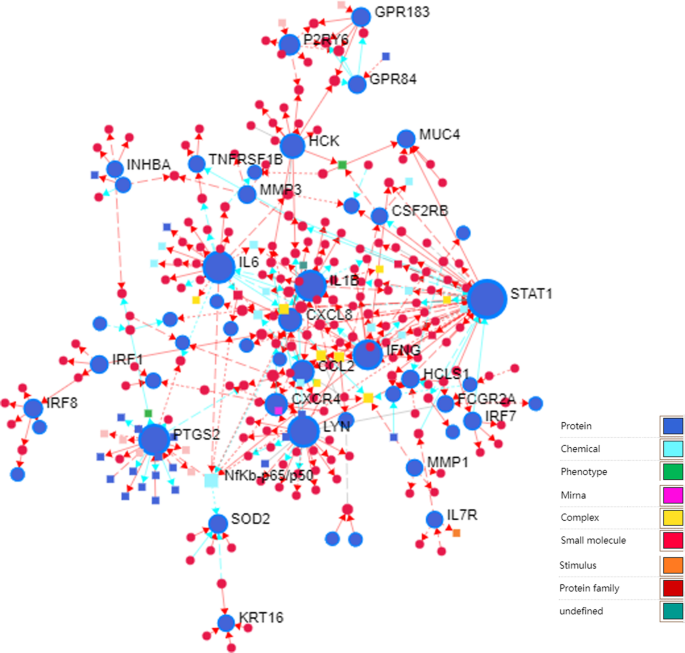
如图所示。5,我们基于SIGNOR2.0, Table构建了DEGs信令信息网络2根据相互作用程度列出了前20位高水平基因,揭示了STAT1、IL6、LYN、PTGS2、IL1B、IFNG、HCK、CXCL8、CCL2、CXCR4等在该网络中广泛参与了化学信号物质的调控。
WGCNA共表达网络及共表达模块的构建
我们进行了网络拓扑分析,以确定WGCNA中相对、平衡的规模独立性和平均连通性的候选功率值。结果,6208个deg(调整P值< 0.05)的RAS样本使用WGCNA算法构建共表达模块。随后,利用flashcluster函数进行分层聚类分析,结果如图所示。6A.软功率阈值β由函数“sft$powerEstimate”确定,如图所示。6B,幂值为6是尺度独立性小于0.8的最小幂值,选择该值生成6208个基因的层次聚类树。最后,基于平均层次聚类和动态树裁剪方法识别出16个模块,每个模块具有不同的颜色和基因。各模块之间具有显著的独立性,计算各模块的特征基因模块值,聚类树如图所示。6C.在所有模块中,绿松石模块的轮毂基因数量最高。然后根据TOM矩阵检测基因模块,分析16个模块之间的相互作用(图5)。6D)。此外,利用本征基因树状图和热图,通过本征基因相关来量化模块相似度(图5)。6E)。
在共表达模块中定义deg
有趣的是,我们发现几乎所有的差异表达基因,特别是上述两个网络分析中的关键节点都涉及到Turquoise模块,包括SOD2、STAT1、PTGS2、IL-6等。因此,我们使用R软件得到的DEGs (log2 (fold-change) | > 1.5和P< 0.05),共表达的677个基因中有254个DEGs。
GSEA富集分析
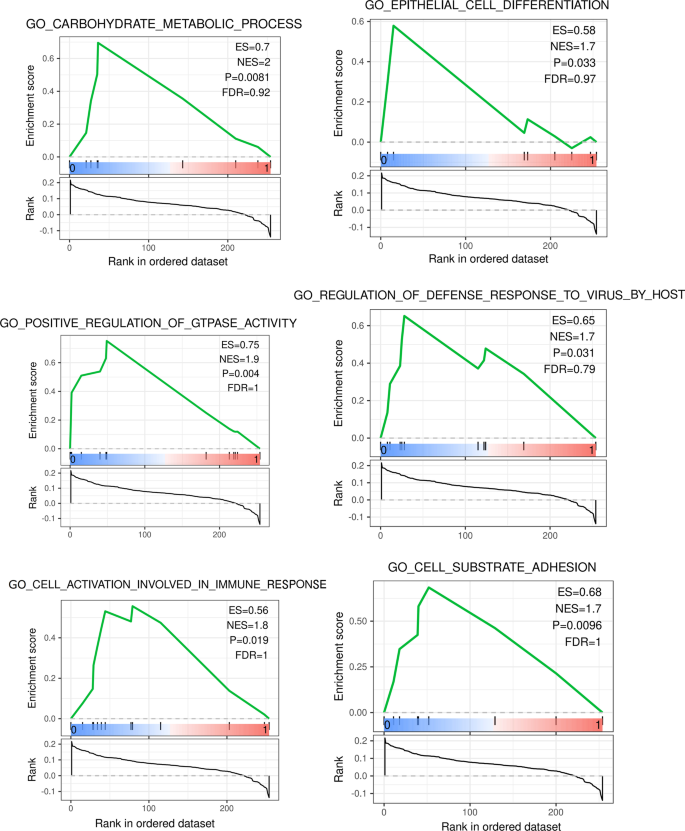
无花果。7给出了基于Go(生物过程)的GSEA富集分析结果。如图所示。8Turquoise模块中DEGs的通路主要集中在免疫反应、病毒感染、上皮细胞、信号转导等方面,其中与RAS高度相关的通路主要包括GTPase活性的正调控、参与免疫反应的T细胞活化、上皮细胞分化、细胞器组织的正调控、细胞底物粘附、宿主对病毒的防御反应的调控、钙介导的信号转导的调控、白细胞介素1的产生等。共富集56个核心靶点,包括ICAM1、CCR7、IL1B、PLEK、CCL4、NCKAP1L、GPR65、ZC3H12A、P2RY6、CCL8、RGS1、CCL2、ARHGAP9、ADAP2、RGS18、ITGAL、LCP1、LILRB1、STAT1、MSN, CORO1A参与2条以上GO通路,其中参与5条通路频率最高的ICAM1。
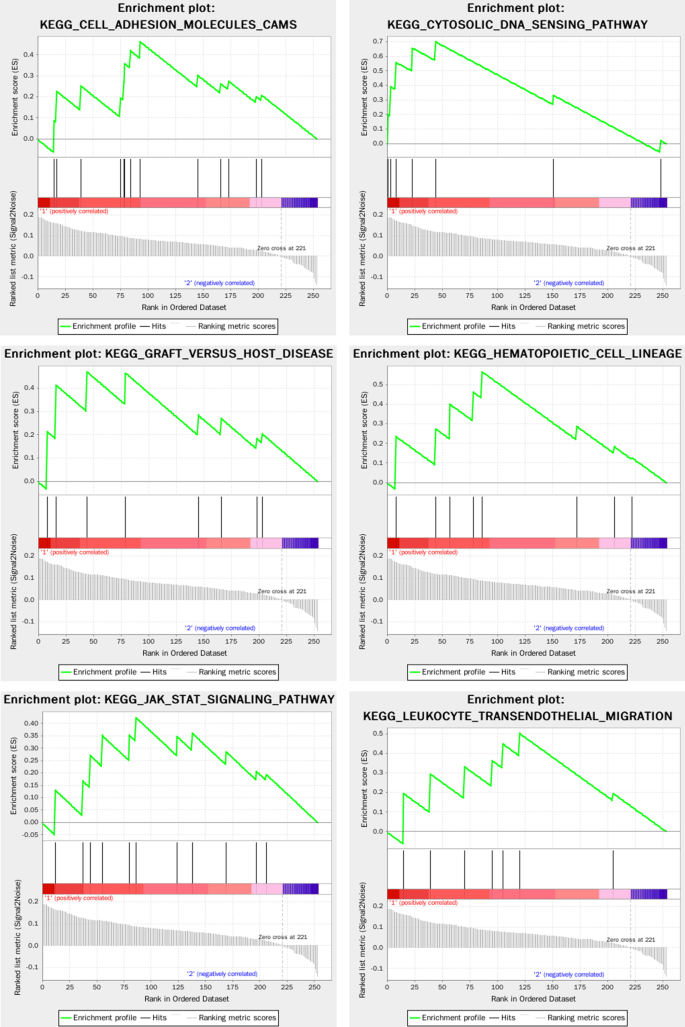
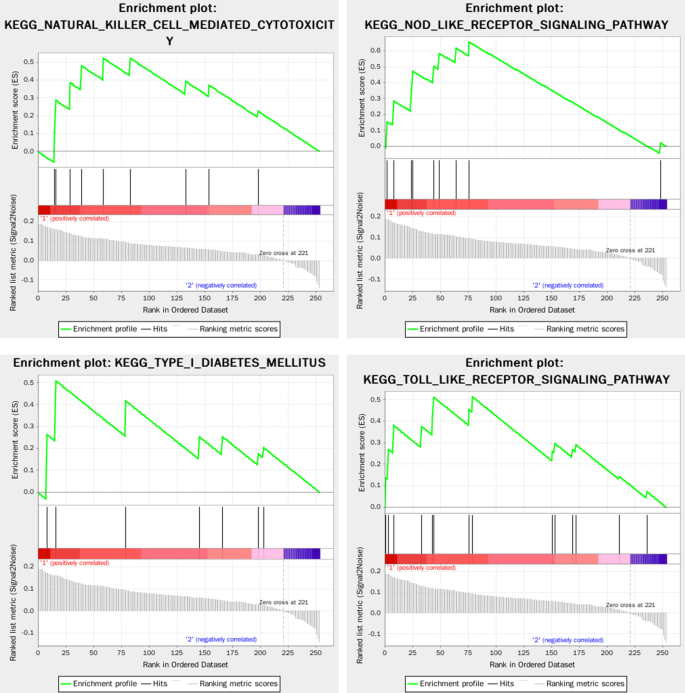
无花果。9给出了基于KEGG的GSEA富集分析结果。与复发RAS高度相关的Turquoise模块中DEGs的通路主要包括细胞粘附分子cams、胞质DNA感应通路、自然杀伤细胞介导的细胞毒性、I型糖尿病、nod样受体信号通路、造血细胞谱系、移植物抗宿主病、白细胞跨内皮迁移、JAK/STAT信号通路、TOLL样受体信号通路等。共富集了55个关键基因,包括IL1B、IL6、CD86、HLA-B、ICAM1、ITGAL、HLA-DMA、HLA-F、HLA-DMB、GZMB、CD2、CCL4、CXCL10、IRF7、IL18、CXCL8、IL7R、CD14、CSF3R, STAT1参与2条以上通路,其中IL1B和IL6靶标参与6条通路频率最高。
枢纽基因的鉴定与验证
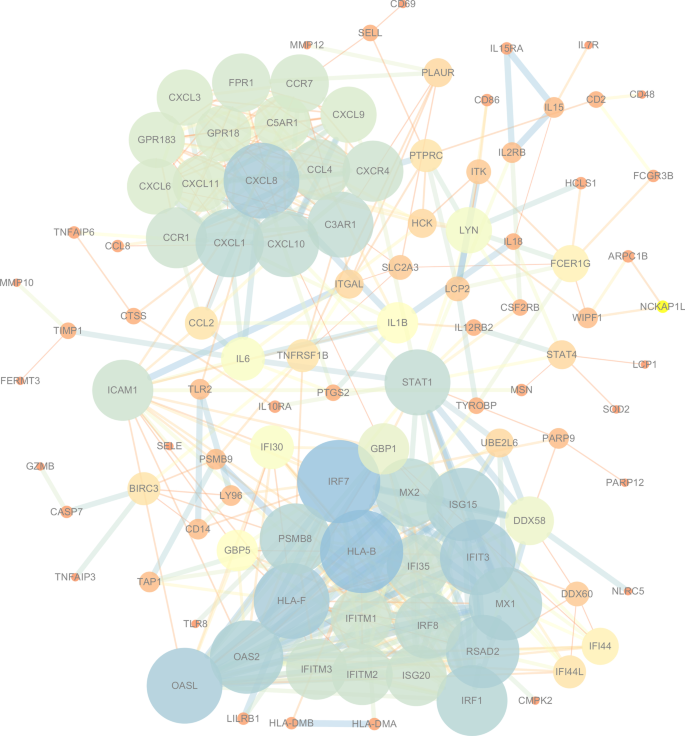
通过GSEA富集分析,我们将Turquoise模块相关通路中涉及的DEGs合并后,基于String数据库构建了富集DEGs的蛋白-蛋白相互作用(protein-protein interaction, PPI)网络(图5)。10).表格3.列出排名前20位的deg的网络参数,如HLA-B、IRF7、HLA-F、IFIT3、osl、CXCL8、OAS2、MX1、ISG15、RSAD2、IRF1等。
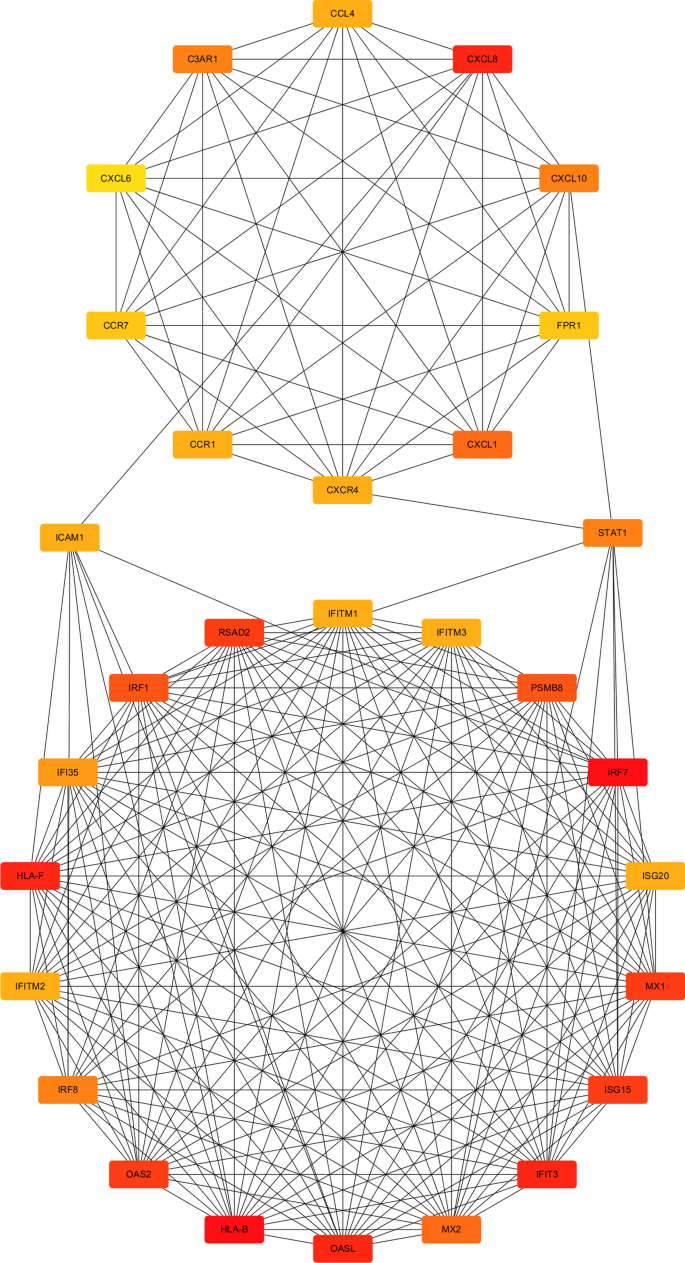
如图所示。11, MCODE模块确定了网络中的两个集群(高度互联区域),分别为17.647和10。蛋白质-蛋白质相互作用网络中的簇通常是蛋白质复合物和通路的一部分,它们在不同类型的网络中有不同的含义。有趣的是,簇1和簇2由STAT1和ICAM1连接。目前的研究表明,ICAM1是STAT1的下游基因,STAT1的激活可诱导ICAM1的表达[25].根据表中的评分参数4,同时结合我们之前构建的信号信息网络和mirna -基因相互作用网络的综合分析,推测STAT1可能是影响RAS过程的关键基因。
筛选潜在的天然化合物
最终根据tccmsp数据库筛选出与STAT1具有高亲和力的8种天然成分,包括染料木素、大豆苷元、山奈酚、白藜芦醇、迷思香酸、雷公藤甲素、槲皮素和(-)-表没食子儿茶素-3-没食子酸酯。表格4显示了上述成分的药代动力学特征。
基于Pharmmapper和富集分析预测潜在靶标
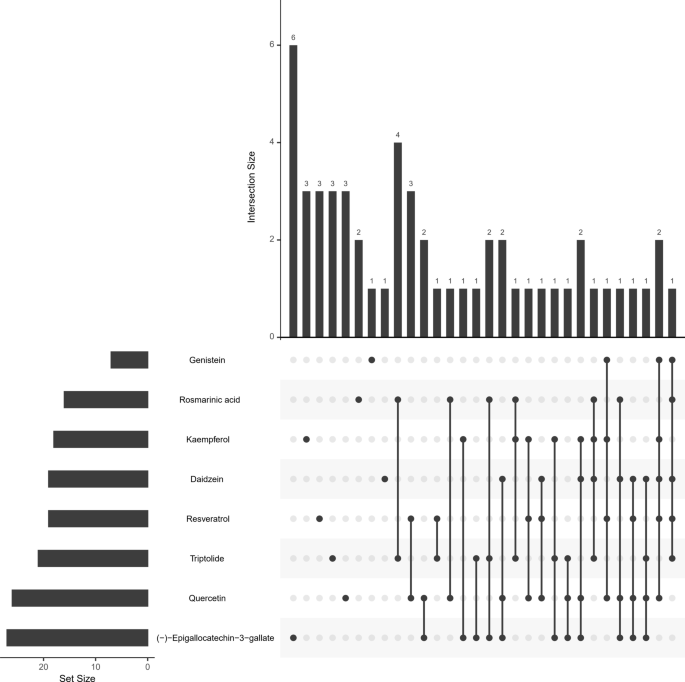
我们在Pharmmapper上预测了上述8种化合物的潜在靶点。表5为RAS预测靶点与deg的交集,图。12表明这些化合物影响RAS的潜在靶点非常相似,也提示这些化合物可能通过多个靶点影响RAS的过程。
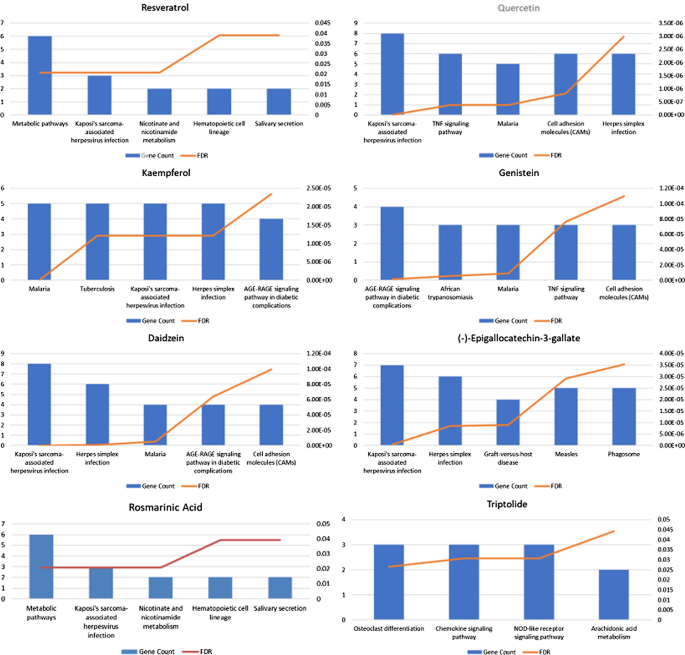
数字13显示了这些化合物预测靶点的KEGG富集分析结果,提示这些化合物影响RAS过程的潜在靶点主要集中在各种病毒感染相关的免疫通路、TNF通路、细胞粘附等生物通路。
确定基于反向对接技术的RAS目标
如图所示。14,反向分子对接的结果表明,不同的化合物可能会结合到STAT1蛋白的不同部位,而具有相似分子结构的化合物则会结合到STAT1蛋白的相似位置。对接分数越高,与蛋白质的结合力越强。从高到低依次为:雷公藤甲素(−9.1 kcal/mol)、(-)-表没食子儿茶素-3-没食子酸酯(−8.1 kcal/mol)、迷思香酸(−7.3 kcal/mol)、槲皮素(−7.3 kcal/mol)、genistein(−7.1 kcal/mol)、大豆黄酮(−6.9 kcal/mol)、山奈酚(−6.9 kcal/mol)、白藜芦醇(−5.6 kcal/mol)。
讨论
在本文中,我们通过差异分析,初步筛选出RAS患者溃疡组织和正常组织中12,548个差异表达基因,最终根据筛选标准将187个基因纳入研究。在构建了这些基因的mirna -基因相互作用网络和信号信息网络后,我们确定了候选关键节点靶点。然后我们将WGCNA应用到所有基因的聚类分析中,发现我们感兴趣的关键节点目标都在同一个模块中,因此我们对该模块中的差异基因进行了进一步的富集分析,并进一步筛选出枢纽基因。然后对感兴趣模块中的hub基因进行功能分析,发现这些hub基因主要参与免疫应答、病毒感染、上皮细胞、信号转导等相关通路。识别了PPI网络,并识别了通过STAT1连接的两个模块。最终确定STAT1介导的多种生物学通路可能影响RAS疾病的过程。推测STAT1可能成为RAS治疗的潜在靶点。最后,利用分子反向对接技术筛选出了几种可能作用于STAT1蛋白的化合物。其中一些化合物已被证实是STAT1蛋白的抑制剂,它们有望成为RAS的潜在治疗药物。
许多因素已经涉及RAS的促进和/或恶化。然而,RAS的主要病因尚不清楚。大量的研究关注于阐明RAS的原因,已经提出了几种可能的致病因素。潜在的病因包括局部和全身条件、阳性家族史、基因易感RAS个体的创伤(在RAS患者中已发现某些基因特异性HLAs)、营养因素(如叶酸和b族维生素缺乏)、免疫因素、社会心理压力和对饮食成分的过敏、局部创伤、营养缺乏、食物过敏、戒烟和心理压力。以及感染性微生物因素等[2,3.,6].
在过去的20年里,大量的研究主要集中在免疫因素上,但很明显,RAS的免疫发病机制没有统一的理论[3.].RAS病因研究的大部分内容,除了在淋巴细胞亚群中的缺陷之外,还包括T淋巴细胞和单核细胞对口腔上皮的细胞毒性作用、免疫复合物血管炎、抗体依赖性细胞介导的细胞毒性,这些研究表明了少量免疫介导反应以及RAS的发展之间存在共同的联系。各种免疫反应可导致口腔上皮细胞内免疫复合物的沉积而引起损伤。然而,引发这些反应的原因还不太明确[2,4,7].研究发现RAS的严重程度与RAS病变中CD4+和CD8+细胞的异常规模、CD4+:CD8+比率的变化、白细胞介素2、干扰素γ水平的升高以及肿瘤坏死因子-α (TNF-α) mRNA的表达有关[3.,4,5,6].在RAS患者外周血单个核细胞中发现大量分泌TNF-α,这标志着TNF在RAS发病机制方面发挥着不可或缺的作用。因此,TNF-α介导的内皮细胞粘附和中性粒细胞趋化作用是炎症过程级联的启动器,从而导致溃疡[5].TNF-α的绝大多数是为了响应toll样受体(TLRs)的激发而产生的,TLRs是一系列与上皮屏障相关和保护的功能性膜受体,不仅具有促进作用,而且具有抗炎作用[4].由于血清免疫球蛋白和自然杀伤细胞水平在RAS患者的正常有限范围内发挥着重要作用,人们开始关注一种有利于T细胞亚群积累的失调的局部细胞介导免疫反应[6].局部免疫反应导致最终的组织抑制表现为RAS。
我们使用crytoscape 3.7.3中的MCODE模块来识别富集差异表达基因中的两个核心Cluster模块。如图所示。11簇1和簇2由STAT1和ICAM1连接,而ICAM1是STAT1的下游蛋白,STAT1的磷酸化程度会影响ICAM1蛋白的表达[26].不难发现,最初影响RAS发病的因素主要集中在干扰素途径中,包括干扰素调节因子(IRF)、干扰素基因启动子和干扰素刺激反应基因(ISG),或涉及病毒感染引起的一种抗病毒蛋白如IFITMs、OAS2或OASL,也被干扰素途径激活。此外,Cluster1还包含RAS患者特异性表达基因,如HLA-B或HLA-F。簇2主要包含趋化因子及其受体。我们认为干扰素途径通过STAT1蛋白激活趋化因子及其受体,已证实基质金属蛋白酶系统与趋化因子网络之间存在串扰,趋化因子及其受体可能调控基质金属蛋白酶活性[28,29],可能影响口腔上皮胶原蛋白的合成和降解,最终表现为溃疡的形式。根据之前的研究和该芯片中的DEGs, RAS中许多类型的基质金属蛋白(MMPs)或金属蛋白酶组织抑制剂(TIMPs)已被证实与正常组织相比存在差异表达[30.,31,32].此外,ICAM1还可以介导胶原蛋白的合成和分解,这也需要STAT1的介导。因此,抑制STAT1可能切断干扰素通路中的一些异常,抑制趋化因子活性,进而影响基质金属蛋白酶的相关活性,影响口腔胶原蛋白的合成或分解,也可能是RAS发生的机制之一。
此外,最新研究证实,牙周炎患者血清或唾液中的Galectin和IL-6水平发生了显著变化[33,34],表明这些因素可能与口腔疾病密切相关。有趣的是,Galectin通路也可能介导RAS疾病的进展,这可能是另一种完全不同于STAT1介导的生物学通路。在本研究中,RAS患者的溃疡组织中Galectin-1、Galectin-2和Galectin-3的表达也发生了显著变化。其中,Galectin-1与过度炎症密切相关,主要通过调节T细胞、B细胞、巨噬细胞和粒细胞等免疫细胞,促进免疫耐受,下调先天和适应性免疫反应[35];Galectin-3不仅影响I型胶原的合成,还影响TIMPs和MMPs的活性[36].最值得注意的是RAS患者溃疡组织中IL-6的表达发生了明显变化。IL-6是我们之前构建的信号信息网络和mirna -基因相互作用网络中的关键节点基因。富集分析也显示IL-6参与多种生物途径。同时IL-6也是激活STAT1的关键因素之一[37], STAT1介导的il -6相关生物学通路在RAS进展中的作用也值得进一步深入研究。
最后,我们也证实了STAT1蛋白是RAS潜在的治疗靶点之一,该靶点可用于筛选潜在的治疗化合物。最后从tcsp数据库中筛选出染料木素、大豆苷元、山奈酚、白藜芦醇、迷迭香酸、雷公藤甲素、槲皮素和(-)-表没食子儿茶素-3-没食子酸酯,它们都是STAT-1抑制剂[38,39,40,41,42].有趣的是,其中一些成分,如迷迭香酸、槲皮素、(-)-没食子儿茶素-3-没食子酸酯、白藜芦醇等,已经制成了治疗口腔溃疡的外用制剂,如槲皮素、(-)-没食子儿茶素-3-没食子酸酯、白藜芦醇[43,44,45].分子反向对接的结果表明,除了雷公藤甲素、(-)-表没食子儿茶素-3-没食子酸酯和白藜芦醇外,其他5种结构相似的化合物(类黄酮)与STAT1的结合位置几乎相同,即该位置可能是类黄酮抑制STAT1蛋白的关键位置。
结论
我们在WGCNA的基础上发现了可能有助于RAS诊断和治疗的潜在生物标志物,推测STAT1是潜在的治疗靶点之一。反向分子对接的结果提示我们可以从STAT-1抑制剂中筛选RAS治疗药物。
数据和材料的可用性
本研究中使用或分析的数据集可根据合理要求从通讯作者处获得。
缩写
- 拉:
-
复发性口腔炎
- NCBI:
-
国家生物技术信息中心
- 地理:
-
基因表达综合
- WGCNA:
-
加权基因共表达网络分析
- 汤姆:
-
拓扑重叠矩阵
- STAT1:
-
信号换能器和转录激活器1
- ICAM1:
-
细胞间粘附分子-1
- TCMSP:
-
中药系统药理学数据库与分析平台
- 度:
-
差异表达基因
- GSEA:
-
基因集富集分析
- 走:
-
基因本体论
- KEGG:
-
京都基因基因组百科全书
- PPI:
-
蛋白质相互作用
- MCODE:
-
分子复合体检测
- 主成分分析:
-
主成分分析
- 兆瓦:
-
分子量
- AlogP:
-
测定分子疏水性的关键
- OB:
-
口服生物利用度
- BBB:
-
血脑屏障
- DL:
-
Drug-likeness
- FASA -:
-
所有带部分负电荷的原子的分数水可达表面积
- TPSA:
-
一种描述分子极性的物理化学性质
- RBN:
-
分子柔韧性的描述,允许自由旋转的键的数量
- EPC:
-
边缘渗透组件
- 跨国公司:
-
最大邻域分量
- DMNC:
-
最大邻域分量密度
- 通常:
-
toll样受体
- 肿瘤坏死因子:
-
肿瘤坏死因子
- IRF:
-
干扰素调节因子
- 研究小组:
-
干扰素刺激反应基因
- 基质金属蛋白酶:
-
基质金属蛋白
- TIMPs:
-
金属蛋白酶组织抑制剂
- il - 6:
-
白细胞介素- 6
- HLA:
-
人白细胞抗原
- CD4:
-
分化聚类4
- CD8:
-
分化聚类8
参考文献
- 1.
Saikaly SK, Saikaly TS, Saikaly LE。复发性阿弗他溃疡:潜在原因和新的治疗方法综述。中华皮肤科杂志2018;29(6):542-52。
- 2.
埃德加NR,萨利赫D,米勒RA。复发性口腔炎综述。中华临床美容皮肤病学杂志2017;10(3):26。
- 3.
Ślebioda张志军,张志军,张志军。复发性口腔炎的发病机制及免疫学方面的作用:文献综述。中华免疫学杂志2014;62(3):205-15。
- 4.
Ślebioda Z, Szponar E, Kowalska A.复发性口腔炎:病因的遗传方面。Adv Dermat Allergol/Postȩpy Dermatologii I Alergologii. 2013;30(2):96。
- 5.
Tarakji B, Gazal G, Al-Maweri SA, Azzeghaiby SN, Alaizari N.牙科医生复发性口腔炎诊断和治疗指南。国际口腔健康杂志:JIOH。2015; 7(5): 74。
- 6.
艾伯森,Hedström L, Bergh H.低水平激光治疗复发性口腔炎和疼痛:一项随机对照试验。口腔外科口腔医学口腔病理口腔放射。2014;117(5):590-4。
- 7.
Belenguer-Guallar I, Jiménez-Soriano Y, claramont - lozano A.复发性口腔炎的治疗。文献综述。中华临床经验杂志2014;6(2):e168。
- 8.
Mangul S, Martin LS, Langmead B, Sanchez-Galan JE, Toma I, Hormozdiari F, Pevzner P, Eskin E.生物信息学和开放数据如何促进资源有限的国家和大学的基础科学。中国生物技术学报2019;37(3):324-6。
- 9.
阿特伍德TK,布莱克福德S,布拉达斯MD,戴维斯A,施耐德MV。对不断发展的生物信息学和数据科学培训需求的全球视角。生物信息学报2019;20(2):398-404。
- 10.
裴刚,陈琳,张伟。WGCNA在蛋白质组学和代谢组学数据分析中的应用。方法酶学杂志2017;585:135-158。
- 11.
田铮,何伟,唐杰,廖旭,杨强,吴勇,吴刚。基于WGCNA的乳腺癌重要模块和生物标志物的识别。Onco Targets Ther. 2020;13:6805。
- 12.
基于系统生物学的靶点识别和药物发现的最新进展。中华医学杂志2018;18(20):1737-1744。
- 13.
Katsila T, Spyroulias GA, Patrinos GP, Matsoukas MT.目标识别和药物发现的计算方法。生物技术学报。2016;14:177-84。
- 14.
药物靶点识别的计算方法。应用于:计算机辅助药物设计。新加坡:施普林格. 2020。p . 163 - 185。
- 15.
Karagkouni D,等。DIANA-TarBase v8:长达十年的实验支持mirna -基因相互作用的集合。核酸学报2018;46(D1): D239-45。
- 16.
李晓霞,王晓燕,王晓燕,等。SIGNOR 2.0,信令网络开放资源2.0:2019更新。核酸条例2020;48(D1): D504-D510。
- 17.
Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA, Paulovich A, Pomeroy SL, Golub TR, Lander ES, Mesirov JP。基因集富集分析:一种解释全基因组表达谱的基于知识的方法。中国生物医学工程学报,2005;32(5):369 - 369。
- 18.
兹克拉奇克D,盖博AL,里昂D,荣格A,怀德S,韦尔塔- cepas J,西蒙诺维奇M,东切娃NT,莫里斯JH,博克P,延森LJ。STRING v11:覆盖范围增加的蛋白质-蛋白质关联网络,支持全基因组实验数据集的功能发现。核酸修正案2019;47(D1): D607-13。
- 19.
Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape:生物分子相互作用网络集成模型的软件环境。Genome Res. 2003;13(11): 2498-504。
- 20.
茹娟,李萍,王娟,周伟,李斌,黄超,李萍,郭铮,陶伟,杨勇,徐旭。tcsp:一个从草药中发现药物的系统药理学数据库。化学通报,2014;6(1):13。
- 21.
王鑫,沈勇,王松,李松,张伟,刘旭,赖林,裴杰,李宏。PharmMapper 2017更新:一个具有综合靶标药效团数据库的潜在药物靶标识别web服务器。《核酸学报》2017;45(W1): W356-60。
- 22.
Trott O, Olson AJ。AutoDock Vina:通过新的评分功能,高效优化和多线程,提高对接的速度和准确性。计算化学。2010;31(2):455-61。
- 23.
德拉诺王。PyMOL分子图形系统,2.0版本。Schrödinger LLC. 2002。
- 24.
紫菜T,诺泽尔SE, Benveniste EN。ifn - β介导的il-8表达抑制需要isgf3组件stat1, stat2和irf-9。细胞因子杂志,2008。
- 25.
摘要A11: STAT1介导实验性口腔癌耐药,与增强的抗肿瘤t细胞反应相关。摘要:AACR-AHNS头颈癌会议:通过基础、临床和转化研究优化生存和生活质量;2019年4月29-30日;德克萨斯州奥斯汀,2020年。
- 26.
Yockell-Lelievre J, Spriet C, Cantin P, Malenfant P, Heliot L, De Launoit Y, Audette M. Stat-1与ets-1的功能合作优化icam-1基因转录。生物化学细胞生物学,2009;87(6):905-18。
- 27.
Janowska-Wieczorek A, Marquez LA, Dobrowsky A, Ratajczak MZ, Cabuhat ML.人类骨髓和外周血CD34+细胞对趋化因子的反应中MMP和TIMP的差异产生。中华血液病杂志2000;28(11):1274-85。
- 28.
Robinson SC, Scott KA, Balkwill FR.单核细胞基质金属蛋白酶-9的趋化因子刺激需要内源性TNF-α。中华免疫学杂志2002;32(2):404-12。
- 29.
王晓燕,王晓燕,王晓燕,等。急性髓系白血病中基质金属蛋白酶系统与趋化因子网络的相互作用。当代医学化学2010;17(36):4448-61。
- 30.
Karasneh JA, Bani‐Hani ME, Alkhateeb AM,等。MMP而非TIMP-1基因多态性与复发性口腔炎的相关性。口腔杂志2014;20(7):693-699。
- 31.
康艳,张雅英。MMP-9在复发性阿弗他溃疡中的表达及意义。中华口腔医学杂志。2008;(05):710-713。
- 32.
康云云,张莹,孙莹。MMP-28在RAU、OLP中的表达及意义研究。OLK口腔医学杂志2011;9:5。
- 33.
伊索拉·G, Polizzi A, Alibrandi A,等。galectin‐3水平作为牙周炎期间冠心病风险来源的分析牙周病杂志,2021;56(3):597-605。
- 34.
Isola G, Giudice AL, Polizzi A, Alibrandi A, Murabito P, Indelicato F.牙周炎患者唾液中白细胞介素-6的不同分布:一项横断面研究。Arch Oral biology . 2021;122:104997。
- 35.
Astorgues-Xerri L, Riveiro ME, Tijeras-Raballand A, Serova M, Neuzillet C, Albert S, Raymond E, Faivre S。《癌症治疗杂志》2014;40(2):307-19。
- 36.
de Boer RA, Voors AA, Muntendam P,等。Galectin-3:心力衰竭发展和进展的一种新的中介因子。中华医学会心力衰竭杂志2009;11(9):811-817。
- 37.
Metwally H, Tanaka T, Li S, Parajuli G, Kang S, Hanieh H, Hashimoto S, Chalise JP, Gemechu Y, Standley DM, Kishimoto T非典型STAT1磷酸化扩展其转录活性,促进lps诱导的IL-6和IL-12p40的产生。科学信号。2020;13(624)。
- 38.
Hämäläinen M, Nieminen R, Vuorela P, Heinonen M, Moilanen E.黄酮类化合物的抗炎作用:染料木素,山奈酚,槲皮素和大豆黄酮抑制STAT-1和NF-κB激活,而黄酮,异鼠李素,柚皮素和天竺萸素只抑制NF-κB激活,以及它们对激活巨噬细胞iNOS表达和NO生成的抑制作用。Mediat inflamm。2007; 2007:1-10。
- 39.
Hamed F, Mcdonagh A, Almaghrabi S, Bakri Y, Tazi-Ahnini R.表没食子儿茶素-3没食子酸酯抑制斑秃患者stat-1/jak2/irf-1/hla-dr/hla-b并降低cd8 mkg2d淋巴细胞。国际环境研究与公共卫生。2018;15(12):2882。
- 40.
红琴T, Xinyu L, Heng G, Lanfang X, Yongfang W, shaasha S.雷公藤甲素通过Jak/STAT途径抑制HaCaT角化细胞IFN-γ信号。植物学报,2011;25(11):1678-85。
- 41.
马晨,王莹,董丽,李敏,蔡伟。白藜芦醇通过抑制NF-κB和JAK/STAT信号通路的抗炎作用。生物化学与生物物理学报。2015;47(3):207-13。
- 42.
杨志华,刘建明,李e,李斌,赵维奎,朴基,马济勇。紫苏乙醇提取物对TNF-α/IFN-γ刺激的人角化细胞HaCaT细胞的抗炎作用。中华民族药理学杂志。2018;211:217-23。
- 43.
郭亮,赵志英,白洁,等。槲皮素与载药壳聚糖复合材料的制备与治疗口腔溃疡。Adv Mater Res. 2012; 583:44-48。
- 44.
陈培良、朱淑贞、郭文华、周爱梅、林家杰、谢玉山。表没食子儿茶素-3没食子酸盐抑制口腔癌细胞的侵袭、上皮-间质转化和肿瘤生长。农业食品化学学报,2011;59(8):3836-44。
- 45.
黄斌,陈浩。-没食子表儿茶素-3-没食子酸酯对口腔溃疡基质金属蛋白酶的抑制作用。RSC ad . 2015;5(30): 23758-66。
确认
首先,我要感谢我的导师JING先生,他是一位可敬、负责、机智的学者,在论文写作的各个阶段都给予了我宝贵的指导。如果没有他的谆谆教诲、令人印象深刻的善良和耐心,我是不可能完成论文的。他敏锐而有力的学术观察不仅对我的论文,而且对我今后的学习都有很大的启发。我还要感谢所有帮助我培养基本学术能力的老师们。我也要感谢青岛大学附属医院的老师和同学们,他们非常配合地参与了这次研究。最后但并非最不重要的,我要感谢我所有的朋友,感谢他们的鼓励和支持。
资金
本研究得到“青岛市中医药2021年发展计划”(No. 2021-zyyq22)资助,主要资助语言编辑服务。
作者信息
从属关系
贡献
MC和FJ对研究的构思有贡献;MC和MF检索和启动基因芯片数据挖掘工作;LL和LX对稿件准备的贡献很大;CZ和LW通过建设性的讨论帮助进行分析;XX和WR整理了全文的表格和图片。所有作者阅读并批准了最终稿件。
相应的作者
道德声明
伦理批准和同意参与
根据《赫尔辛基宣言》的伦理指导原则,建立实验方案,并经青岛大学附属医院人类伦理委员会批准。获得个体或监护人的书面知情同意。
发表同意书
不适用。
相互竞争的利益
作者声明没有竞争的非财务/财务利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
权利与权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
曹明,李林,徐林,徐明。et al。STAT1:复发性口腔炎的新候选生物标志物和潜在治疗靶点。口腔健康21日,524(2021)。https://doi.org/10.1186/s12903-021-01776-w
收到了:
接受:
发表:
关键字
- 复发性口腔炎
- WGCNA
- 中心的基因
- STAT1
- 反向分子对接