抽象的
人的口腔中含有一个复杂的微生物群落,直到最近,一直没有得到很好的特点。已经利用分子工具研究开始枚举和量化居住在口腔内的各种龛的品种;然而,几乎所有的研究已经揭示了额外的新物种,而鲜为人知的是,口腔微生物群落或者它如何与疾病变化的结构动力学。在口腔范围高达700种细菌多样性的当前估计,尽管在任何单个个体这个数字低得多。口服微生物是负责常见的慢性疾病,并且建议是全身性的人类疾病的哨兵。微阵列现在被用来研究口腔菌群以系统和稳健的方式。虽然这项技术还比较年轻,改进已在技术的所有方面,包括提供从非特异性完美匹配杂交之间更好地歧视进步使(密切相关)杂交。本文综述使用基于凝胶的微阵列和初步整合该技术到需要复杂的微生物群落结构的全系统的研究和口腔诊断设备发展的一个单一的设备中的核心技术。
介绍
微生物包括每个人的生物系统的主要部分。它们是皮肤,肠道和口腔/咽系统的正常居民。大多数情况下,我们付出很少考虑到居住在我们的身体的微生物物种的众人。只有当这种关系上不去,导致如炎症或其他疾病状态不良的生理反应,我们充分意识到他们的存在。口腔疾病,龋齿,牙周炎和是最常见的慢性预防人类疾病中,并且与他们的环境复合微生物相互作用,其它微生物的结果,和主机[1].除了微生物发病机制之外,越来越多的证据表明,微生物口腔植物群可以作为人体系统性疾病的哨兵,如糖尿病,心脏病,低孕重的分娩和肺炎[2-15].我们研究团队进行研究和技术开发计划的一个基本前提是,如果不了解内源性微生物群的关系,对人体作为一个系统的描述是不完整的。
尽管有些物种与口腔疾病有关(例如。变形链球菌是龋齿的重要贡献者Porphyromonas gingivalis到牙周病),我们对可能有更复杂的微生物病因学的疾病的了解非常不全面。基于分子普查研究的估计表明,人类口腔是数百种独特微生物的家园[16-23].这些种群分布在牙齿、组织表面和唾液中。值得注意的是,大多数这些微生物还没有被带入纯培养[16那17那24-28] - 表征他们的生理和可能的致病因素必不可少的前奏。虽然分子的调查显示很多关于存在于口腔中的物种,很少有人知道在任何一个单独的微生物群落动态(通过时间或改变不良饮食和健康状况),或如何口腔菌群谁拥有个人内变化特定疾病或易患疾病。因此,我们发展社区结构的更全面的了解在口腔内,从而提供了互动的微生物种群之间的表征及人类宿主的重要基础是必不可少的。
这篇简要的综述了用于快速和可靠的口腔微生物群鉴定的DNA微阵列技术的发展。潜在的前提是,这些信息将在解剖与进展性疾病(如携带和牙周炎)相关的复杂微生物病原学方面有很大的用处。我们正在开发的诊断设备的首要目标是在小唾液样本中提供关键微生物种群的快速识别和近似定量。因此,本报告的主要重点是为唾液诊断设备的发展提供概念和技术背景,旨在快速测量唾液的微生物组成。
微阵列技术的前景与实践
微阵列已被证明可用于高度复合的分析,如全基因组表达研究,并越来越多地应用于复杂微生物群落的研究。通过小样本与单个阵列杂交,可以快速检测数百到数千个目标序列(例如,对应于不同的微生物种群)。微生物群落分析有几种探针设计策略;我们更倾向于设计探针来检测核糖体RNA(rRNA),以检测在每个系统发育等级上共享的序列的共同区域,从而接近分类等级(例如物种、属、门和域)。这些系统发育微阵列已被用于在包括唾液在内的多种环境中鉴定细菌物种[29-34].用于rRNA微生物识别的最广泛使用的微阵列是印刷在玻璃载玻片上的平面阵列[29-32那34那35],虽然也有玻璃以外的表面[36那37].
我们的系统发育阵列采用凝胶垫技术,在装载寡核苷酸探针之前,由100 μm × 100 μm × 20 μm的小聚丙烯酰胺垫组成的有序阵列被光聚合到位[38那39].垫尺寸可以尺寸变化,取决于蚀刻到光刻掩模中的图案。凝胶垫阵列和其他微阵列表面之间的根本差异是各种聚合物凝胶元件产生高密度的三维“试管”阵列。探针与聚合物骨架而不是固体衬底共价交联,该固定化探针浓度能够在单个凝胶元件内达到10mm [39].凝胶垫微阵列的解决方案性质具有许多理论和实用益处;在开发口头诊断的背景下,我们看到凝胶垫阵列平台的几个主要优势:1)它们是可重用的,从而减少了用户的阵列到阵列可变性和成本;2)它们可以直接检测天然放大的rRNA,缓解在酶促扩增期间发生的偏差[28那40],从而为目标丰度提供更直接的衡量标准;3)它们具有较高的探针固定能力,有助于检测低丰度目标(特别是在没有扩增步骤的情况下)。
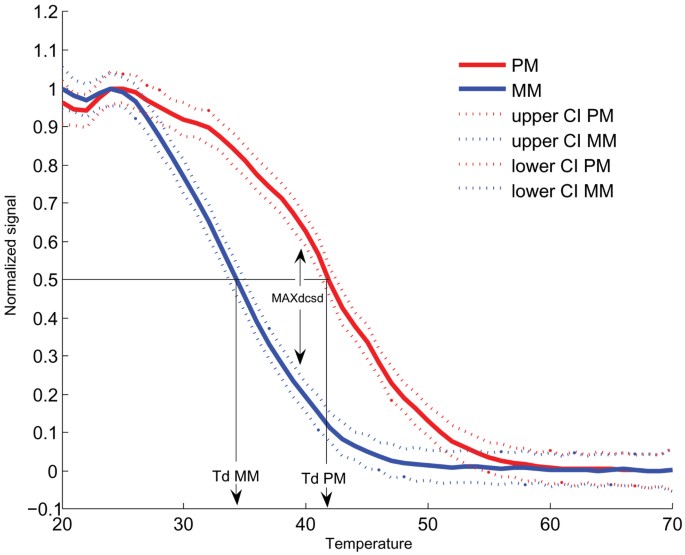
所有微阵列实验的挑战包括我们自己的,正在确认样品中的物种的身份,具有高度置信度。为了检测非特异性杂交,研究人员通常使用使用错配探针,其相对于每个完美匹配探针依次依次不同。另一种策略是对每个生物使用多种探针[31].我们的方法利用凝胶元件的溶液相性质,并使用离解非平衡分析来分辨阵列上的完美和不完美双晶。一个阅读器(显微镜和相机)和一个热平台结合使用来描述目标从每个探针的分离,通过测量残余信号,随着温度的增加。由此产生的熔化剖面(信号与温度)被用作双相成分的测量(见图)2).这种分离方法已被证明有助于从密切相关的非目标序列中区分目标,因为包含一个或多个错配的目标比完全匹配的目标更早地分离[33那39那41-45].Discriminating perfect-match from mismatch hybridizations – a key step toward determining the presence or absence of a particular target (e.g. species) – is also influenced by such factors as the diffusivity of the gel array, the quality of the target material, and image analysis methods.
提高凝胶阵列的扩散率
如上所述,凝胶焊盘技术具有许多积极的属性。然而,聚丙烯酰胺凝胶和凝胶焊盘制造方法确实对探针靶相互作用的其他溶液相行为施加了某些限制。如图所示1,垫倾向于在三维凝胶元件的外围和边缘产生更强烈的信号,而不是在整个凝胶元件中产生统一的信号。造成这种现象的原因是多方面的。当然,凝胶元素有一个确定的孔隙大小,限制了凝胶内部的进入;根据我们的经验,目标分子范围从20-150个核苷酸是首选的,并导致最强和均匀的信号强度在单个垫。其次,非常高的固定化探针浓度对杂化动力学和平衡有出乎意料的影响,Livshits等人雄辩地描述并详细描述为延迟扩散理论[46那47].为了说明,当目标首先找到探测时,目标将与它相关联在焊盘的最可访问部分 - 表面上。然后通过用不同的探针分子重复解离和重新分配的过程,目标“针”本身进入垫的内部。当探针的浓度高时,该过程较慢,因为靶标的探针与靶向相互作用。该延迟扩散的该过程决定了在凝胶垫中建立平衡的时间,其中靶的最大数量是均匀的约束。对于微生物群落分析和明确识别从阵列的不完美双工的完美来说是重要的,即由于迟钝的扩散而实现最终平衡所需的时间比实验杂交条件长。在执行“熔体”实验时,反向情况也适用。理论迅速碰撞实践,因为系统发育阵列的极少用户有奢侈等待建立均衡关联和解离条件,以识别完美和不完美的双工。因此,我们研究了使用非平衡解离以区分完美匹配和错配杂交,现在正在开发微流体技术以加速反应动力学。
众所周知,主动混合或流动可显著提高微阵列性能,导致绝对信号强度增加和低背景或非特异性结合[48-50].连续的洗涤(或流动)促进了所有目标的分离,但它对错配目标的影响比完美匹配目标更明显,因为错配目标的分离速率常数高于完美匹配目标的分离速率常数。因此,使用连续流系统可以增强对完美匹配和错配杂交的区分。为了在我们的微阵列平台上实现这一点,我们使用了微流控设备,它非常适合引入洗涤协议。由于附在机械泵上的流控线,初始微流控系统需要大的缓冲体积(200~400 μL)。然而,随着技术的进步,我们设想嵌入或集成一个包含机械泵系统的小模块,以减少体积。
凝胶元件制造工艺的改进可能使凝胶内部更容易接近和“溶液状”,减轻上述延迟扩散。Rubina等人[51例如,描述了一种用于凝胶元件阵列制造的共聚技术,其消除了凝胶元件上的光刻掩模和相关的“边缘”。捕获探针与源板中的聚合物预先混合,用常规机器人排列,并在适当的地方光聚合,产生“凝胶液”。由于在聚合前均匀地分布捕获探针,因此聚合后它们同样均匀地分布在整个凝胶体积中[51那52)(图1,面板b)。在另一个进步中,将凝胶元件解剖到数百个微柱(Selamat等,未发表)中,从而改善了凝胶元素内的探针分布(图1,小组C)。与普通凝胶元件相比,这种“华夫格状”凝胶元件可用于探针固定的有效表面积增加了三倍。然而,固定化探针的均匀分布并不能消除延迟扩散。凝胶孔隙率、聚合物材料和固定化探针浓度也对整个三维凝胶元件的重复缔合/解离产生深远影响[53].为了对抗这些影响,一些新的可调谐聚合物正在开发和测试中,其中一些可以产生高达300纳米的孔隙,或平均细菌大小的1/3。与用于凝胶垫的原始聚丙烯酰胺配方相比,杂交动力学在50分子靶上的速度至少是前者的两倍。这些聚合物中的一些已经提高了热稳定性,这是快速热熔实验和实现平衡结合条件(在杂交和洗涤过程中)比以前更快的特性。上述的微柱改性也增强了长目标分子在凝胶元件中的扩散能力。与普通凝胶元件相比,这种修饰可以提高杂交率和信号强度达5倍。杂交动力学的这些改进可能会提高微生物检测研究中信号检测(如假阴性和假阳性信号)的准确性(Hong等人,未发表)。结合微流体装置的主动流动,凝胶元件阵列的新发展有望实现口腔微生物群落组成的快速分析。
样品材料的信号增加
标本制备和处理中的几个步骤可能会影响微阵列结果。例如,环境样本中细菌种类裂解的不同效率会使微阵列结果产生偏差[34].为此,我们已经优化了从唾液样本微生物捕获和裂解方案。此外,目标分子的长度影响的材料的扩散率。由于尺寸和的16S rRNA(近1500个碱基)的高度结构化的性质,我们rRNA基因片段,以确保有效渗透到微阵列的凝胶元件。在该步骤的碎片协议优化确保质量的杂交和可重复的结果[54那55].
样品制备的最后一步是用荧光团标记目标材料。工业标准,如Cy3,经常是现成的,但正如最近报道的,不一定是需要高温的实验的最佳选择[56].例如,Cy3和罗丹明红在20°C和80°C之间分别损失高达80%和60%的荧光强度。因此,对于使用这些和类似荧光团的解离实验,信号强度的降低是目标的解离和荧光的损失的结合。如果没有质量控制,例如在凝胶阵列中添加标记有与目标相同荧光团的控制探针,就很难解旋这两个过程。因此,非温度依赖性荧光染料是首选的信号检测在较高的温度用于解离分析。标记目标材料时需要考虑的另一个特点是使用末端标记还是内部标记策略。信号强度可能随荧光团的位置而变化,特别是当它附着在目标分子的末端或附近时[57]. 随机标记内部碱基的策略有助于减少信号强度的这些变化。此外,它们具有多次标记目标分子的能力,这可能进一步提高信号强度。
提高图像分析
鉴于我们需要在实验过程中对数组进行成像,我们需要能够将相同的网格应用到整个图像集(或堆栈)的软件。在实验过程中,一些人为因素,如不对齐的网格、阵列上的颗粒物质、斑点overshine或气泡形成,都可能对数据质量和结果熔化剖面产生不利影响[58-60].我们小组使用的标准图像分析工具不保留图像,考虑到其开发时的计算和存储限制,并且不可能进行额外的再分析[43].在过去的一年中,已经开发了几个新的软件程序(Labarray,AMIA和ISTACKX),允许用户从具有显着质量控制措施的图像中提取信号强度。Labarray是一个图像采集和分析工具,允许实时监控探头 - 双工解离,并且可以立即量化在特定温度下拍摄的每个图像内的所有斑点的强度[61].每个图像都保存,可以通过Labarray或其他图像分析软件重新分析。使用LabVIEW(National Instruments,Austin TX)开发的Labarray可以同时控制其他仪器组件。MATLAB和ISTACKX的自动微阵列图像分析(AMIA)工具箱是分析工具,允许用户分析由其他图像采集程序收集的一系列图像[58](克里克等人。,未发表的)。AMIA提供了许多统计和视觉工具,使用户能够定量评估图像分析,包括用于超出寄存器的图像的“线程”容量(从使用运动控制器)。ISTACKX程序具有一个电影功能,同时显示所提出的网格放置,以确保网格的准确性。
微阵列的解释
完全匹配与失配杂交的鉴别
一旦分析了图像并且处理了所处理的信号强度(包括背景减法和归一化),就被解释为评估它们是否来自完美匹配或不匹配目标。评估单点沿解离曲线(例如,初始信号强度和T.D.(当温度为初始信号强度的50%时))是一个有用的数据简化步骤,可以简化数据处理。T的比较D.完美匹配探头的值通常大于TD.不匹配探针与预期的目标相同,给定更大的完美匹配稳定性[43那56那62,但并不是所有的研究都报告了使用TD.[42].这个结果并不意外为TD.受许多变量,如长度和目标,位置浓度和错配的类型和扩散速率的影响。因为序列和靶的浓度是未知的口腔粘膜样本,对T依赖于D.独自一人是有问题的。解离曲线上的其他点显示出比T更好的分辨力D..维克等人描述了一种新的度量,叫做Tdw,测量的温度K.D.(缔合常数)在离解速率曲线上达到最大值[63].虽然还有待观察哪些参数影响Tdw,它确实优于TD.区分完全匹配和不匹配探针。因此,这个新参数在完美匹配/不匹配分析中可能是一个有用的工具。此外,Urakawa等人利用判别指标和神经网络来表征曲线间最优判别区域;然而,这些指标并没有提供曲线的统计比较[42].Bugli等人开发了一个功能性的方差分析计算器,该计算器应用统计工具来比较整个分解曲线的差异,并计算一个新的度量,MAXDCSD.最大限度DCSD是一条分解曲线的95%置信区间下限与另一条曲线的95%置信区间上限之间归一化信号强度的最大差值(Bugli等人,已提交)(见图2).在应用功能方差分析计算器从环境样本核酸,MAXDCSD当TD.没有(Eyers等人提交),并且,与Tdw,最大值的温度DCSD发生也可能是要监视的有用参数。
物种鉴定
各种测量参数的集成需要复杂的计算过程。例如,神经网络可以处理许多可以定义每个探测目标熔化型材的不同参数。在初步微阵列的研究中,具有来自人口腔中发现的微生物的已知靶序列的15,584探针靶序列,用神经网络鉴定了85%的预测的完美匹配探针 - 目标双工;但是,分析还产生了几个假阳性读数[59]. 为了突出神经网络的效用,用于测试神经网络的数据包罗万象,表明它能够在次优条件下表现良好。Urisman等人采用了另一种计算方法进行物种鉴定[64].他们的计算方法将在单一温度下观测到的信号强度与预测的能量分布进行比较,从而得出一个相似分数[64].为了解释他们的数据集的相似性分数,他们开发了参数来计算给定相似性分数的被检测物种的概率。因为他们的系统复杂性很低(每个样本不超过两种病毒),要使这种方法适应于其他系统,特别是来自复杂群落的系统,将需要额外的优化和标准化,可能还需要多次迭代来识别。尽管如此,该方法可以应用于所有的微阵列,平面和多维,并将其应用于口腔样本的杂交结果将是有趣的。本文所描述的分析和技术进步的应用有望进一步提高我们区分完美匹配和错配杂交事件的能力。随着神经网络和其他计算方法的强大分辨力,口腔微生物群落的快速、样本-答案诊断正在成为现实。
结论
除了今天技术成熟的地平线之外,在其环境中实时监测微生物的承诺以及成本效益的诊断,将允许早期检测和预防医疗干预。以前,微阵列辅助在2002年出现后不久的快速鉴定SARS的致病剂,并且作为人类疾病诊断被引入微阵列[65那66].技术集成是这一进步的关键,而通过微流控技术实现的规模经济和独特的理化特性对于提高微阵列输出的特异性和敏感性至关重要。事实上,我们正在构建的集成微流控微阵列设备允许在实验设备中监测杂交和分离的动力学。此外,将标本制备与微流控-微阵列设备集成在同一微流控卡上,将使研究和临床社区从繁琐和费力的方法中解脱出来。随着我们向完全集成设备的方向发展,外部和内部卡上质量控制标准正在开发中,以使该设备适合于护理点诊断。已有研究将微生物的反应与各种生理参数和疾病发展联系起来[67-69].然而,最终,将微流控微阵列设备与复杂的生物信息学联系起来,将使微生物群和人类健康的纵向和横向研究成为可能,这在目前是不可想象的。对内源性微生物群与人体的关系有了更全面的认识;这些研究将帮助我们识别人类疾病(口腔和全身)的诊断标记物,可以用来设计有效的干预策略。
参考文献
- 1.
改变牙周病的治疗模式:临床意义。中华医学会牙周病防治杂志。2000,20(6):513 - 513。
- 2.
牙周炎:冠心病的危险因素?牙周病杂志。1998,3:127-141。
- 3.
关键词:心血管疾病,牙周病,全局性炎症,比较研究临床诊断实验室免疫。2002,9:425-432。10.1128 / cdli.9.2.425 - 432.2002。
- 4.
Kinane DF,罗威GD:如何牙周疾病可能导致心血管疾病。牙周炎。2000,23:121-126。10.1034 / j.1600-0757.2000.2230112.x。
- 5.
Scannapieco FA, Papandonatos GD, Dunford RG:全国抽样调查人群中口腔条件和呼吸道疾病之间的关联。牙周病杂志。1998,3:251-256。
- 6.
Terpenning MS, Taylor GW, Lopatin DE, Kerr CK, Dominguez BL, Loesche WJ:吸入性肺炎:老年退伍军人的牙科和口腔危险因素。中华老年医学杂志。2001,49:557-563。10.1046 / j.1532-5415.2001.49113.x。
- 7.
Miller Ls,Manwell Ma,Newbold D,Reding Me,Rasheed A,Blodgett J,Kornman Ks:减少牙周炎和糖尿病控制之间的关系:报告9例。j渡晨醇。1992,63:843-848。
- 8.
类风湿关节炎与牙周炎的关系。牙周病杂志,2001,12:779-787。10.1902 / jop.2001.72.6.779。
- 9.
Offenbacher S, Jared HL, O'Reilly PG, Wells SR, Salvi GE, Lawrence HP, Socransky SS, Beck JD:牙周炎相关妊娠并发症的潜在致病机制。牙周病杂志。1998,3:233-250。
- 10.
Offenbacher S, Lieff S, Boggess KA, Murtha AP, Madianos PN, Champagne CM, McKaig RG, Jared HL, Mauriello SM, Auten RL等:母亲牙周炎与早产儿。第一部分:早产和生长受限的产科结局。牙周病杂志。2001,6:164-174。10.1902 / annals.2001.6.1.164。
- 11.
Madianos PN、Lieff S、Murtha AP、Boggess KA、Auten RL、Beck JD、Ofenbacher S:母亲牙周炎和早产。第二部分:母体感染和胎儿暴露。安牙周病。2001,6:175-182.10.1902/annals.2001.6.1.175。
- 12.
格罗西SG,发电公司RJ:牙周病和糖尿病:一个双向的关系。安Periodontol。1998年,3:51-61。
- 13.
全身性疾病的牙周表现。中国科学(d辑:地球科学),2001,46(2):12 - 14。
- 14.
Scannapieco FA:美国牙周病学会立场论文:牙周病是系统性疾病的潜在危险因素。牙周病杂志。1998,9:841-850。
- 15.
Teng YT, Taylor GW, scannapeco F, Kinane DF, Curtis M, Beck JD, Kogon S:牙周健康与系统性疾病。中国兽医学报,2002,27(6):457 - 461。
- 16.
Aas JA, Paster BJ, Stokes LN, Olsen I, Dewhirst FE:口腔正常菌群的定义。中国临床微生物学杂志。2005,43(4):521 - 532。10.1128 / jcm.43.11.5721 - 5732.2005。
- 17.
Paster BJ, Boches SK, Galvin JL, Ericson RE, Lau CN, Levanos VA, Sahasrabudhe A, Dewhirst FE:人类龈下菌斑的细菌多样性。中国微生物学杂志,2003,19(4):427 - 434。10.1128 / jb.183.12.3770 - 3783.2001。
- 18.
口腔微生物群落:生物膜、相互作用和遗传系统。微生物学进展。2000,54:413-437。10.1146 / annurev.micro.54.1.413。
- 19.
Corby PM, Lyons-Weiler J, Bretz WA, Hart TC, Aas JA, Boumenna T, Goss J, Corby AL, Junior HM, Weyant RJ,等:儿童早期龋的微生物风险指标。中国临床微生物学杂志。2005,43(4):553 - 559。10.1128 / jcm.43.11.5753 - 5759.2005。
- 20.
李勇,顾青云,徐静,徐军。基于pcr变性梯度凝胶电泳的口腔微生物多样性研究。中国海洋大学学报(自然科学版),2005,34(4):531 - 534。
- 21.
Sakamoto M,Takeuchi Y,Umeda M,Ishikawa I,Benno Y:终端RFLP分析的应用在健康受试者唾液中的口腔细菌植物和牙周炎患者的应用。J med microbiol。2003,52:79-89。10.1099 / jmm.0.04991-0。
- 22.
Dymock D,Weightman A,Scully C,Wade W:与齿槽脓肿相关的微生物区系的分子分析。临床微生物学杂志。1996, 34: 537-542.
- 23.
摩尔我们,Moore Lv:牙周病的细菌。课厅2000. 1994年,5:66-77。
- 24.
人类龈下裂隙内的细菌多样性。《美国国家科学院学报》上。1999年,96:14547 - 14552。10.1073 / pnas.96.25.14547。
- 25.
Munson MA, Banerjee A, Watson TF, Wade WG:龋齿相关微生物群的分子分析。临床微生物学杂志。2004,42:3023-3029。10.1128 / jcm.42.7.3023 - 3029.2004。
- 26.
Becker Mr,Past BJ,Leys EJ,Moeschberger Ml,Kenyon SG,Galvin JL,Boches SK,Dewhirst Fe,Griffen Al:与儿童龋齿相关的细菌物种的分子分析。J Clar Microbiol。2002,40:1001-1009。10.1128 / JCM.40.3.1001-1009.2002。
- 27.
Sakamoto M, Umeda M, Ishikawa I, Benno Y:通过16S rDNA文库序列分析比较一名健康受试者和两名牙周炎患者唾液中的口腔菌群。微生物免疫学报。2000,44:643-652。
- 28.
de Lillo A, Ashley FP, Palmer RM, Munson MA, Kyriacou L, Weightman AJ, Wade WG:使用多种通用聚合酶链反应引物检测新的牙龈下细菌系统类型。口腔微生物学杂志。2006,21:61-68。
- 29.
Small J, Call DR, Brockman FJ, Straub TM, Chandler DP:利用寡核苷酸芯片直接检测土壤提取物中的16S rRNA。App Enviro Microbio. 2001, 67: 4708-4716。10.1128 / aem.67.10.4708 - 4716.2001。
- 30.
Loy A,Lehner A,Lee N,Adamczyk J,Meier H,Ernst J,Schleifer K-H,Wagner M:基于16S rRNA基因的寡核苷酸微阵列检测环境中所有公认的硫酸盐还原原核生物谱系。应用环境微生物学。2002年,68:5064-5081.10.1128/AEM.68.10.5064-5081.2002。
- 31.
Wilson KH,Wilson WJ,Radosevich JL,DeSantis TZ,Viswanathan VS,Kuczmarski TA,Andersen GL:小亚单位核糖体DNA探针的高密度微阵列。应用环境微生物学。2002,68:2535-2541.10.1128/AEM.68.5.2535-2541.2002。
- 32.
Palmer C, Bik EM, Eisen MB, Eckburg PB, Sana TR, Wolber PK, Relman DA, Brown PO:复杂微生物种群的快速定量分析。核酸学报2006,34:e5-10.1093/nar/gnj007。
- 33.
小泉,Kelly JJ, Nakagawa T, Urakawa H, El-Fantroussi S, Al-Muzaini S, Fukui M, Urushigawa Y, Stahl DA:利用pcr -变性梯度凝胶电泳、RNA-DNA膜杂交和DNA芯片技术并行表征厌氧降解甲苯和乙苯的微生物群。环境微生物学杂志,2002,29(4):531 - 534。10.1128 / aem.68.7.3215 - 3225.2002。
- 34.
Desantis TZ, Stone CE, Murray SR, Moberg JP, Andersen GL:利用微阵列快速定量和分类来自原核和真核起源的环境DNA。微生物微生物学杂志,2005,34(4):421 - 427。10.1016 / j.femsle.2005.03.016。
- 35.
在平面寡核苷酸微阵列上直接检测16S rRNA的序列与结构应用环境微生物学报,2003,29(4):429 - 434。10.1128 / aem.69.5.2950 - 2958.2003。
- 36.
Rudi K,Skulberg OM,Skulberg R,Jakobsen KS:序列特异性标记的16S rRNA基因寡核苷酸探针在通过阵列杂交进行蓝藻丰度和多样性遗传分析中的应用。应用环境微生物学。2000年,66:4004-4011.10.1128/AEM.66.9.4004-4011.2000。
- 37.
Socransky SS,史密斯C,马丁L,帕斯特BJ,德惠斯特FE,莱AE: “棋盘” DNA-DNA杂交。生物学技术。1994年,17:788-792。
- 38.
Guschin D, Mobarry B, Proudnikov D, Stahl D, Rittmann B, Mirzabekov A:微生物学中作为基因传感器的寡核苷酸芯片。应用环境微生物学学报,1997,13(2):337 - 342。
- 39.
Yershov G,巴斯基V,Belgovskiy A,基里洛夫E,Kreindlin E,Ivanov的I,Parinov S,Guschin d,Drobishev A,Dubiley S,等人:上的寡核苷酸的微芯片的DNA分析和诊断。《美国国家科学院学报》上。1996年,93:4913-4918。10.1073 / pnas.93.10.4913。
- 40.
Acinas SG, Sarma-Rupavtarm R, Klepac-Ceraj V, Polz MF: pcr诱导的序列伪伪和偏差:来自从同一样本构建的两个16S rRNA克隆库的比较的观察。应用环境微生物学。2005,31:8966-8969。10.1128 / aem.71.12.8966 - 8969.2005。
- 41.
刘文涛,Mirzabekov AD, Stahl DA:用于微生物鉴定研究的寡核苷酸芯片的优化:非平衡解离方法。环境微生物学报,2001,3:619-29。10.1046 / j.1462-2920.2001.00233.x。
- 42.
浦河H,埃尔Fantroussi S,Smidt H,斯穆特JC,Tribou EH,凯利JJ,诺布尔PA,斯塔尔DA:在寡核苷酸微阵列的单碱基对错配歧视的优化。应用环境微生物学。2003,69:2848年至2856年。10.1128 / AEM.69.5.2848-2856.2003。
- 43.
乌拉克川H,Noble Pa,El Fantroussi S,Kelly JJ,Stahl DA:使用寡核苷酸微阵列和神经网络分析来单键对终端失配的判断。应用环境微生物学。2002,68:235-244。10.1128 / AEM.68.1.235-244.2002。
- 44.
El Fantroussi S, Urakawa H, Bernhard AE, Kelly JJ, Noble PA, Smidt H, Yershov GM, Stahl DA:通过与寡核苷酸微阵列杂交的天然rrna的热解离分析,直接分析环境微生物群体。应用环境微生物学报,2003,29(6):593 - 598。10.1128 / aem.69.4.2377 - 2382.2003。
- 45.
Fotin A, Drobyshev A, Proudnikov D, Perov A, Mirzabekov A:寡脱氧核糖核酸芯片上双链的平行热力学分析。核酸学报1998,26:1515-1521。10.1093 / nar / 26.6.1515。
- 46.
DNA与凝胶固定化寡核苷酸杂交动力学的理论分析。acta physologica sinica(生物科学学报),32(6):593 - 598。
- 47.
Livshits MA, Ivanov IB, Mirzabekov AD, Florent'ev VL: [DNA测序与一个寡核苷酸矩阵(SHOM)杂交。杂交后DNA洗脱理论]。杂志(Mosk)。1992年,26日:1298 - 1313。
- 48.
Kohara Y:通过湍流在毛细血管中排列的珠子排列的DNA探针的杂交反应动力学。肛门化学。2003,75:3079-3085。10.1021 / AC0341214。
- 49.
拜纳姆·马,戈登·GB:使用微流体行星离心混合增强杂交。肛门化学。2004年,76:7039-7044.10.1021/ac048840+。
- 50.
Vanderhoeven J, Pappaert K, Dutta B, Vanhummelen P, Baron GV, Desmet G:利用小型化技术增强DNA微阵列。电泳。2004,25:3677-3686。10.1002 / elps.200406116。
- 51.
鲁比娜AY,Pan'kov SV,娃EI,Pen'kov DN,Butygin AV,Vasiliskov VA,丘季诺夫AV,Mikheikin AL,米哈伊VM,米扎贝科夫AD:水凝胶液滴的微芯片与固定的DNA:属性和方法用于大规模生产。肛门生物化学。2004年,325:92-106。10.1016 / j.ab.2003.10.010。
- 52.
Rubina AY, Dementieva EI, Stomakhin AA, Darii EL, Pan'kov SV, Barsky VE, Ivanov SM, Konovalova EV, Mirzabekov AD:基于水凝胶的蛋白质微芯片:制造,性能和应用。生物技术。2003,34:1008-1022。
- 53.
Sorokin NV,Chechetkin VR,Livshits MA,Pan'kov SV,Donnikov MY,Gryadunov DA,Lapa SA,Zadestatelev AS:用寡核苷酸凝胶微芯片区分完全和不匹配的双链体:杂交过程中热力学和动力学效应的作用。生物分子结构动力学杂志。2005, 22: 725-734.
- 54.
Mehlmann M, Townsend MB, Stears RL, Kuchta RD, Rowlen KL:病毒RNA微阵列分析碎片化条件的优化。生物化学学报,2005,32(4):427 - 434。10.1016 / j.ab.2005.09.036。
- 55.
Kelly JJ, Chernov BK, Tovstanovsky I, Mirzabekov AD, Bavykin SG:生成自由基的配合物作为快速有效碎片化和芯片杂交核酸荧光标记的工具。生物化学学报。2002,31(4):413 - 418。10.1016 / s0003 - 2697(02) 00388 - 3。
- 56.
Liu W-T, Wu J-H, Li ES- y, Selamat ES:荧光标记的温度变化及其对DNA芯片研究的影响。应用微生物学学报,2005,31(4):453- 457。10.1128 / aem.71.10.6453 - 6457.2005。
- 57.
Zhang L, Hurek T, Reinhold-Hurek B:荧光标记的位置是决定微阵列杂交信号强度的关键因素。acta physica sinica, 2005, 33: e166-10.1093/nar/gni156。
- 58.
White AM,Daly DS,Willse AR,Protic M,Chandler DP:MATLAB自动微阵列图像分析工具箱。生物信息学。2005,21:3578-3579.10.1093/bioinformatics/bti576。
- 59.
Pozhitkov A, Chernov B, Yershov G, Noble PA:用人工神经网络评价凝胶垫寡核苷酸芯片技术。环境科学学报。2005,31(6):863 - 876。10.1128 / aem.71.12.8663 - 8676.2005。
- 60。
Li X,Gu W,Mohan S,Baylink DJ:DNA微阵列:他们的使用和误用。微循环。2002,9:13-22。10.1038 / sj.mn.7800118。
- 61.
吴jk - k,刘w -t: LabArray:微阵列实时成像分析工具。生物信息学。2005,21:689-690。10.1093 /生物信息学/ bti088。
- 62.
李易西,吴JKK,吴建华,刘文泰:用非平衡解离方法评价平面寡核苷酸芯片的单碱基对识别能力。环境微生物学。2004,6:1197-1202.10.1111/j.1462-2920.2004.00648.x。
- 63.
芯片非平衡解离曲线和解离速率常数作为评估寡核苷酸探针特异性的方法。核酸学报2006,34:e26-10.1093/nar/gnj024。
- 64.
Wang D ., DeRisi J: E-Predict:基于DNA芯片杂交模式的物种鉴定方法。生物谷bioon . 2005, 6: R78-10.1186/gb-2005-6-9-r78。
- 65.
王德,乌斯马坦A,刘yt,springer m,ksiazek tg,erdman dd,mardis er,hickenbotham m,magrini v,Eldred J等:使用DNA微阵列进行病毒发现和序列恢复。Plos Biol。2003,1:e2-10.1371 / journal.pbio.0000002。
- 66.
Wu L,Williams PM,Koch WH:基于微阵列的诊断试验的临床应用。生物技术。2005, 39: 577-582.
- 67.
牙周病原体的风险指标。牙周病杂志,1998,19(4):513 - 513。
- 68.
吸烟与牙龈下菌群的关系。临床牙周病杂志,2001,28(4):427 - 434。10.1034 / j.1600 - 051 x.2001.028005377.x。
- 69.
Haffajee Ad,Smith C,Torresyap G,Thompson M,Guerrero D,Socransky SS:手动和动力牙刷的疗效(II)。对微生物参数的影响。J Clin Suchontol。2001,28:947-954。10.1034 / J.1600-051X.2001.028010947.x。
确认
这项工作得到了国家卫生研究院(NIH)/NIDCR (U01 DE14955)对DAS、LMS和EMLS的资助;NIH (5R01AI059517)至DPC;NASA (MSMT-2004-0045-0066)到DAS、JCS和HHL;美国DARPA (DABT63-99-1-0009)与DAS和JCS;新加坡国立大学(R-288-000-008-112);
作者信息
从属关系
相应的作者
额外的信息
利益争夺
作者声明他们没有相互竞争的利益。
作者的贡献
所有作者阅读并批准了最终的手稿。
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
斯塔克,E.M.L.,斯穆特,J.C.,斯穆特,L.M。等等。探讨口腔健康与口腔微生物群落关系的技术进展BMC口腔健康6,S10(2006年)。https://doi.org/10.1186/1472-6831-6-S1-S10
关键词
- 复杂的微生物群落
- 口腔微生物群
- 人类口腔
- 失配探针
- 微生物群落配置文件